Raskere analyse av DNA-mangfoldet i mudder og tarm

Når Robertsen analyserer DNA er det ikke enkeltorganismer som analyseres, men gigantiske samlinger av DNA fra tusenvis av bakterier samlet på ett sted.
Forskere søker på de mest ugjestmilde steder verden over etter nye forbindelser som for eksempel kan utvikles til medisiner, ha potensiale i industriell virksomhet eller gi ny kunnskap om verden rundt oss. Denne søken og analysen av DNA materiale hos en samling av organismer kalles Metagenomics.
Mudder og tarm
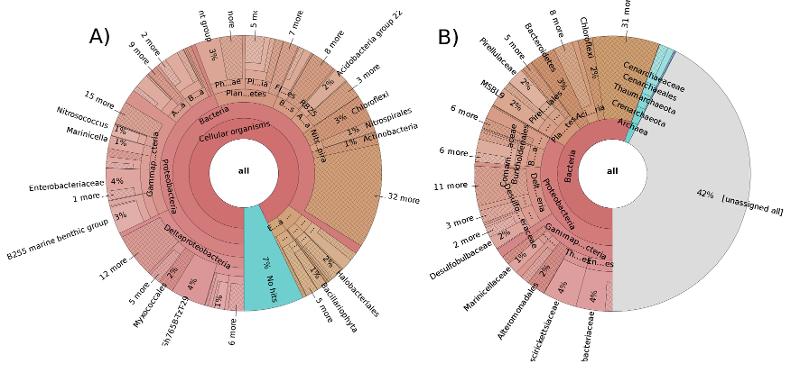
– Nå er det hverken mudder- eller tarmbakterier som er min største forskningsinteresse, smiler Robertsen, men fra disse to kildene finnes det tilgjengelige metagenom og dermed store mengder data jeg kunne bruke under utviklingen av et nytt verktøy for DNA-analyse. Det mange forskere som jobber med marin DNA prospektering ved UiT, så metagenomet som er hentet fra havbunnen i Hinlopenstredet i Barentshavet er av interesse i vårt fagmiljø, mens metagenomet fra en elg-bæsj funnet i Grunnfjorden, er derimot litt mer tilfeldig valgt kan du si.
Det ligger i genene
Den universelle koden for liv, DNA, avleses nå med høyere hastighet enn noen gang påpeker han. Med nye instrumenter og teknikker det blitt mulig å finne informasjon om DNA fra en hel samling mikroorganismer i en neve gjørme fra havets bunn og ikke lengre bare informasjon om èn bakterie om gangen. Men om innsamlingen er uspesifikk, vil forskerne likevel finne ut helt spesifikt hva alt DNA de har funnet kan kode for av interessante egenskaper.
Mangler kapasitet
– Tidligere kunne et søk i DNA banken gjøres på en ordinær PC, men størrelse på databankene øker dramatisk etter hvert som verktøyene for å lese av DNA tråden har blitt mer effektive, det adderes store mengder DNA koder til internasjonale databanker hver eneste dag, forteller han.
Det har etter hvert oppstått store utfordringer knyttet til kapasitet og effektivitet i disse databankene og forskerne trenger nye verktøy for å klare å håndtere datamengdene.
Og det er her målet med Robertsen sitt arbeid kommer inn, nemlig å lage et brukervennlig og effektivt analyseverktøy som kan håndtere store datasett på kortest mulig tid.
Nytt analyseverktøy
– Vi har utviklet META-pipe, som er et gratis og lett tilgjengelig verktøy for analyse av DNA prøver. Vårt fokus har hovedsakelig vært marine metagenom, men det vil kunne brukes til DNA analyser fra mange kilder. Dette systemet tilbyr en fullverdig analyse og ivaretar behovet for preprosessering, taxonomi og kartlegging av funksjon.
Ifølge Robertsen kan META-pipe bruke regnekapasitet fra supercomputere som Stallo, i kombinasjon med andre nasjonale nettverk av maskiner, blant annet NelS, Norstore og Norsk sekvenseringssenter. Forskningsgruppen som Robertsen er en del av samarbeider med forskere i Finland og Tsjekkia, og deres dataressurser har også vært tilgjengelige i utviklingen av META-pipe.
Informatikeren fullførte en doktorgrad på temaet 19. mai 2017. Han var tilknyttet forskerskolen BioStruct-National graduate school in structural biology og prosjektet var en del av Nasjonalt senter for strukturbiologi - NorStruct, ved Institutt for kjemi, UiT. Han ble veiledet av Nils-Peder Willassen, Institutt for kjemi, Lars Ailo Bongo, Institutt for informatikk og Peik Haugen, Institutt for kjemi, alle fra UiT-Norges arktiske universitet. Han fortsetter nå i forskningsgruppen med en 3-årig post doc.
Doktorgradsavhandling: META-pipe – Distributed Pipeline Analysis of Marine Metagenomic Sequence Data.