Vi kan lykkes, men det krever modige grep fra helsemyndighetene og private finansieringskilder.
Nyhetsarkiv
20.11.2024:
Kronikk: Hurtigdiagnostikk er nødvendig for å vinne kampen mot antibiotikaresistens
Uten rask diagnostikk blir antibiotikabehandlingen i de første timene og dagene kvalifisert gjetting. Vi må få opp farten, ellers vinner bakteriene krigen. Det skriver forskerne i RESPNOR-prosjektet på Haukeland i en kronikk i Dagens Medisin.
28.10.2024:
Superbakterien som truer sykehusene (forskersonen.no)
Se for deg en bakterie som kan utvikle forsvarsvåpen mot medisinene våre og som også kan dele disse våpnene med andre bakteriearter. Møt superbakterien Klebsiella pneumoniae

20.09.2024:
Kronikk: Hva bør Norge bidra med for å redusere antibiotika-krisen?
Utviklingen av nye antibiotika dekker ikke dagens behov, og er fullstendig utilstrekkelig for å dekke behovet vi vet kommer. For å stille forberedt må vi få fart på forskningen og tenke nytt om antibiotikamarkedet.
28.08.2024:
Finner alle superbakteriene med ny teknikk
Forskere ved Universitetet i Oslo har vært med på å utvikle en ny metode, som gir oversikt over flere antibiotikaresistente bakterier samtidig - ikke bare en av gangen. Det kan være et viktig steg for å hindre spredning av alvorlige infeksjoner på sykehus.
07.08.2024:
Pakistan unngår superbakterier fra Vesten (uio.no)
Forskning viser at antibiotikaresistente bakteriestammer som er vanlige i Norge, Storbritannia og USA ikke har fått fotfeste i Pakistan, til tross for utbredt bruk av antibiotika i denne regionen.

10.06.2024:
Slik forsvarer E. coli seg mot antibiotika (Gemini.no)
AMR BRIDGE-nytt: Når bakterien oppdager skader på arvestoffet, sender den ut et SOS-signal som gjør at forsvaret prioriteres.

11.03.2024:
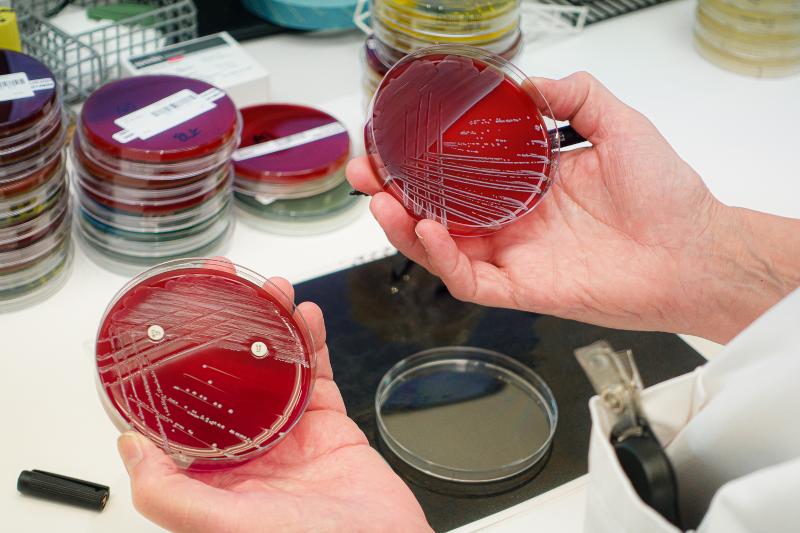
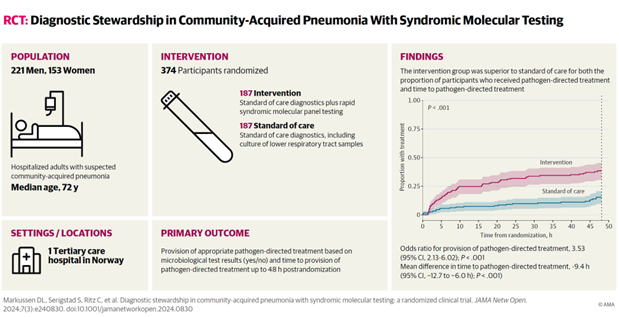
Lungebetennelse: Hurtigtesting gir raskere og mer målrettet behandling
Ny forskning fra Universitetet i Bergen viser at hurtigtesting av pasienter i akuttmottaket kan gi bedre og mer målrettet behandling av samfunnservervet lungebetennelse. På verdensbasis er dette en av de vanligste årsakene til sykehusinnleggelser og dødsfall.
12.01.2024:
Noen antibiotika gir mer multiresistente og sykdomsfremkallende E. coli
Etter å ha analysert 5000 E. coli-infeksjoner fra Norge og Storbritannia i perioden 2001-2017 er resultatene entydige. Det er en klar årsakssammenheng mellom bruk av antibiotikatypen cephalosporiner og økt forekomst av den multiresistente E. coli-klonen ST131-C2.

14.12.2023:
Gemini: Ny metode baner vei for nye antibiotika
Ved NTNU har man nå en lovende kandidat for antibiotika mot resistente gule stafylokokker. Bak funnet ligger metodikk som kan bli viktig i kampen mot antibiotika-resistens.

24.10.2023:
Ghana: Mye Klebsiella-resistens i sykehus, lite ellers i samfunnet
En ny studie viser at det er mye antibiotikaresistent Klebsiella på sykehus, mens omfanget er mye mindre utenfor sykehusene.

13.09.2023:
Ny studie kan bane vei for effektiv behandling av E. coli-infeksjon
Målrettede angrep på E. coli-bakteriens beskyttende kapsel kan bidra til å forebygge og behandle alvorlige infeksjoner i blodet, viser ny studie.
18.09.2023:
Effektive antibiotikatiltak har gitt gode resultater
Forekomsten av antibiotikaresistens her i Norge er fortsatt lav, og salget av antibiotika til landdyr er på sitt laveste nivå på 300 år.
09.03.2022:
Kreftmedisin kan gi antibiotikaresistens (forskning.no)
En mye brukt kreftmedisin kan gjøre bakterier motstandsdyktige mot antibiotika i laboratoriet, viser en norsk studie. Men mer forskning må til før vi vet om det samme skjer i pasienter.
06.12.2021:
Spydspiss i kampen mot antibiotikaresistens (pingvinavisa.no)
Rundt oss vokser antallet med antibiotikaresistente bakterier. Dette er bakterier som lærer seg å tåle alt, og som det ikke finnes noen kur mot. Men i fagmiljøene jobbes det godt, blant annet på laboratoriet til Nasjonal kompetansetjeneste for påvisning av antibiotikaresistens (K-res) ved UNN Tromsø.
14.05.2021:
Forskere vet nå mer om hvordan antibiotikaresistens utvikler seg i Norge (forskning.no)
Økningen av antibiotikaresistens blant E. coli er koblet til spesifikke varianter av bakterien, viser en ny studie.
10.03.2021:
Avdekker historien bak sykehusbakterier
Jukka Corander og hans team ved UiO publiserer sykehusbakterienes historie i Nature - den er lengre enn du kanskje tror. Corander leder et av prosjektene i Trond Mohn stiftelse og Tromsø forskningsstiftelse sitt AMR-program.
For mere informasjon, klikke her.
For hele publikasjonen i Nature, klikk her.