Energiberegninger forklarer biologisk evolusjon
 |
| Nybakt doktor Geir Isaksen (tredje fra venstre) omkranset av komiteen som bedømte oppgaven hans; Ronny Helland fra UiT, Natalie Reuter fra UiB og nobelprisvinner i kjemi fra 2013, Arieh Warshel, University of Southern California, USA. Foto: Vibeke Os |
Naturen har over tid funnet løsninger på hvordan biologiske prosesser tilpasses miljø og temperatur. Slik er det også for enzymer som er naturens spesialdesignede super-katalysatorer og som driver livsnødvendige kjemiske reaksjoner i kroppen. Isaksen har jobbet med å kartlegge hvordan tarmenzymet trypsin fra laks er finjustert for å kunne fungere ved den lave temperaturen i havet, i tillegg til å utvikle verktøy for databeregninger av komplekse biologiske prosesser.
 |
| Tallknuseren Stallo, har vært svært viktig i de beregningene som Isaksen har utført. Supercomputeren som er en av de største i Norden er installert i det nye Teknologibygget der overskuddsvarmen fra maskinen varmer opp bygget. Foto: Thilo Bubek |
Videreføring av en nobelprisvinners arbeid
Grunnlaget for de datamodellene som Isaksen har brukt i sitt arbeid ble lagt av nobelprisvinner i kjemi fra 2013, Arieh Warshel, som også var førsteopponent under disputasen. Warshel var en pioner på området og blant de første som tok i bruk datamaskiner for å beregne kjemiske reaksjoner. Nå er såkalte teoretiske beregninger en forutsetning for blant annet kreftforskning og utvikling av medisiner.
Evolusjon på atomnivå
Kjemiske reaksjoner går saktere i kulde og trypsin fra laks må kompensere for dette på et eller annet vis.
-Vi har forsøkt å avsløre hvordan naturen har funnet løsninger på molekylært nivå, slik at vi selv kan designe molekyler som er tilpasset spesielle oppgaver. For å kunne beregne komplekse reaksjoner på atom-nivå er datamodeller uunnværlige, forklarer Isaksen
Energi forklarer alt
Datamodellen som Isaksen har brukt baserer seg på å finne hvert enkelt atom sitt individuelle energibidrag til en biologiske prosess og der fysikkens lover er bestemmende for hvilke bindinger som kan dannes eller brytes. Dersom en reaksjon ikke kan forklares med riktig energibalanse så vil den heller ikke finne sted.
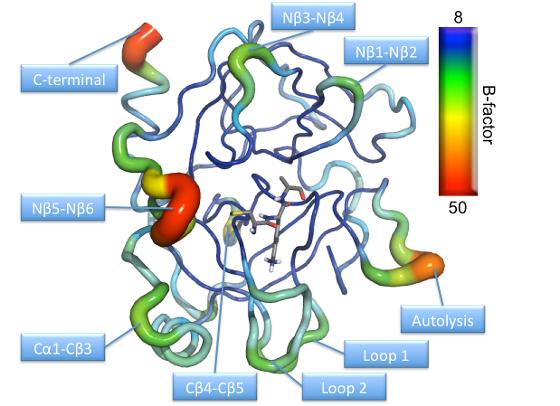 |
| Modell av tarmenzymet trypsin fra laks. Fargene i modellen angir hvilke deler av enzymet som er lite bevegelig (blå) og hvilke deler som er løsere og beveger seg mer (rød). Illustrasjon: Geir Villy Isaksen |
Lagde dataprogram for kjemiske beregninger
-Jeg har vært opptatt av å utvikle et dataprogram, "Qgui", som synliggjør hvilke parametere og betingelser som ligger i bunnen av de beregninger vi gjør. Mye brukte programmer som "Maestro" fungerer på en slik måte at man bare kan laste inn data og hente ut resultater og tallmateriale på andre siden uten nødvendigvis å ha kjennskap til kjemien bak. Da er det fort gjort å miste kontrollen på hvilke parametere og koder som ligger i bunnen av beregningene, utdyper Isaksen.
Ifølge Isaksen kan "Qgui" simulere virkeligheten og visualisere resultatet av forskernes beregninger. Programmet syr sammen en rekke oppgaver som å forberede forsøk, sette opp forsøk, gjennomføre beregninger og optimalisere reaksjoner. Dataprogrammet har vist seg å effektivisere detaljerte energiberegninger for biologiske systemer, i tillegg til å være mer brukervennlig.
Overflaten viktigst for temperaturtilpasning
Litt overraskende fant han ut at overflaten av trypsinet, som tilsynelatende er langt unna reaksjonssenteret, har mest å si for enzymets effektivitet. Men har tidligere trodd at aminosyrer i, eller nært, reaksjonssenteret har vært viktigst.
-Våre resultater er basert på detaljerte energiberegninger av over 100 000 individuelle datasimuleringer og har resultert i ny og viktig kunnskap om hvordan enzymer fungerer på molekylnivå, og har gitt oss et bedre verktøy for å skreddersy biologiske molekyler, oppsummerer Isaksen.
Tittel på avhandlingen var "Protein Dynamics Regulates Enzyme Enthalpy-Entropy Balance" og ble forsvart 26. juni. Arbeidet var tilknyttet CTCC - Center for Computational Chemistry og veiledet av Bjørn Olav Brandsdal.
Litt spesielt var det at nobelprisvinner i kjemi fra 2013, Arieh Warshel fra Southern California University, var førsteopponent under Geir sin disputas.
Les også:




