Blått - bekrefter at datamodellen fungerer
Kjemiske forsøk på laben kan være både tidkrevende, dyre, risikofylte og har i tillegg en del begrensninger som ikke finnes i en imaginær kjemisk verden, der «alt går an». Maarten Beerepoot har i sitt doktorgradsarbeid jobbet med teoretiske kjemiforsøk og videreutviklet metoder som kan gjøre computerberegninger mer realistiske.
Vil få med mer av virkeligheten i sine kjemiske beregninger
I etablerte metoder for kjemiske beregninger er molekylene «tatt ut av sin sammenheng» og gir en forenklet fremstilling av virkeligheten, og bare ved å addere et par vannmolekyler inn i beregningene kan kompleksiteten øke eksponensielt.
-Virkeligheten er jo mye mer komplisert enn som så, smiler Beerepoot, og vi har forsøkt å finne ut mer om hvordan vi skal skrive inn realistiske omgivelser i de teoretiske forsøkene våre.
-De biologiske prosessene som vi ønsker å beskrive er ofte så kompliserte at heller ikke de største datamaskinene har nok regnekraft for å gjøre dette 100% nøyaktig, men med vår metode forsøker vi å komme så nær virkeligheten som mulig, forklarer han. Man kan jo tenke seg for et kraftig verktøy dette ville vært om vi fra datamaskinen kunne beregne hvordan medisiner oppfører seg i kroppen vår eller hvordan forurensning påvirker plante og dyreliv i naturen.
I vår forskning har vi etablert et modellsystem der vi beskriver krefter som påvirker hverandre inne i et stort protein, dvs at vi har et mer komplekst system enn vannmolekyler, men vi er samtidig et stykke unna samspillet i en menneskecelle.
Tester computermodeller med selvlysende protein
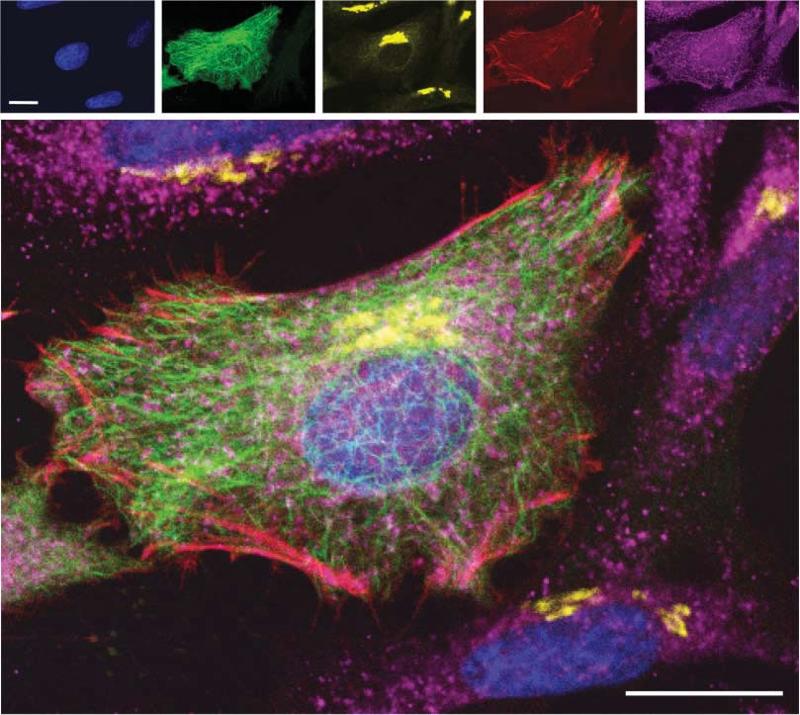
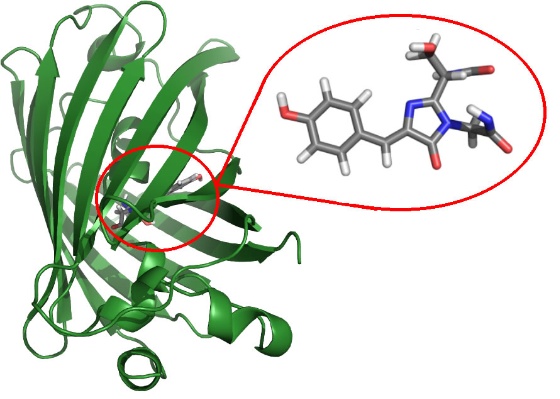
Modellsystemet som har vært benyttet i Beerepoot sitt arbeid er herlig visuelt, nemlig Green fluorescent protein. GFP (Green Fluorescent Protein) kommer opprinnelig fra maneter som lyser i mørket på dyphavet.
Dette selvlysende proteinet har siden dets oppdagelse vært mye brukt som en merkelapp på andre proteiner for å kunne se de «usynlige» prosessene i kroppens celler. GFP brukes blant annet i medisinsk forskning fordi den kraftige selvlysende fargen gjør at stoffer som har fått en GFT-lapp på seg er lette å lokalisere inne i cellene. I mikroskop kan forskerne følge med på om f.eks medisin når frem til riktig «avdeling» i syke celler.
Kjemikeren brukte GFP som modellsystem fordi dette er et stort og komplekst protein og fordi hans beregninger på en enkel måte kunne sammenliknes med eksperimentelle data, f. eks kan små genetiske justeringer i proteinet gjøre proteinet selvlysende blått eller gult. Beerepoot brukte datamodeller for å beregne hva som skulle til for å endre proteinet fra å lyse grønt til å lyse gult/blått, og de samme endringene kunne gjøres i praksis på laboratoriet for å sjekke om proteinet endret farge.

Ifølge Beerepoot har de klart å få godt samsvar mellom databeregningene og de eksperimentelle forsøkene, noe som igjen er et steg videre i prosessen med å utarbeide mer realistiske teoretiske beregninger. De fleste biologiske reaksjoner påvirkes på en aller annen måte av det miljøet de befinner seg i, f.eks det salte havet, den sure magesekken, den oksygenfattige myra, eller i en fettholdig membran i cellene.
-Vi jobber med å utvikle modeller som skal beskrive virkeligheten så nøyaktig som mulig, og må hele tiden overveie kompleksitet og datakraft mot grad av nøyaktighet, avslutter Beerepoot.
Computerbaserte metoder har i 2-3 tiår vært brukt for å kalkulere kjemiske reaksjoner, men ved CTCC – Centre for Theoretical and Computational Chemistry ved UiT er forskerne nå et skritt nærmere realistiske beregninger av mekanismer i store biologiske molekyler.
Beerepoot forsvarte sin avhandling 13. mai 2016, tittel på oppgaven var: «Calculating molecular properties in realistic environments». Han er tilknyttet CTCC - Centre for Theoretical and Computational Chemistry, ved Institutt for kjemi, UiT og forskerskolen BioStruct.
Under doktorgradsarbeidet ble han veiledet av Kenneth Ruud, biveiledere var Bjørn Olav Brandsdal og Luca Frediani, alle fra Centre for Theoretical and Computational Chemistry, Institutt for kjemi, UiT.




